 HOME | ÍNDICE POR TÍTULO | NORMAS PUBLICACIÓN
HOME | ÍNDICE POR TÍTULO | NORMAS PUBLICACIÓN Espacios. Vol. 37 (Nº 36) Año 2016. Pág. 21
Alba Lorena BENAVIDES Sierra 1; Daniel Hernando PRIETO Herrera 2; João Hugo MARTINS Trovão 3; Guilherme Fernando RIBEIRO 4; Gustavo de Souza MATIAS 5; Fernando Henrique LERMEN 6; Leocir Rogerio Lopes PIRES 7
Recibido: 04/07/16 • Aprobado: 25/08/2016
RESUMEN: La microbiota de quesos frescos está compuesta por una diversidad de bacterias que incluye especies que se han relacionado con enfermedades alimentarias. El objetivo de este estudio es identificar bacterias potencialmente productoras de aminas biógenas aisladas de queso blanco fresco tipo Guayanés, Guayamano, Mozzarella, Búfala, Cabra y Cuajada elaborados en Venezuela en dos periodos de tiempo. La microbiota identificada fue presuntiva de: Escherichia coli 1, Enterococcus faecalis, Enterococcus faecium, Staphylococcus sciuri, Staphylococcus xylosus, Staphylococcus carnosus, Pseudomonas fluorescens, Burkholderia cepacia y Lactobacillus fructivorans. Se determinó la capacidad para descarboxilar los aminoácidos histidina, tirosina y ornitina a 19 bacterias pertenecientes a 5 grupos de microorganismos. |
ABSTRACT: The microbiota of fresh cheese is made by a variety of bacteria that includes species that have been associated with foodborne illness. The object of this study is to identify potentially biogenic amines producing bacteria isolated from fresh white cheese type Guayanés, Guayamano, Mozzarella, Búfala, Cabra and Cuajada made in Venezuela in two periods of time. The microbiota was identified presumptively of Escherichia coli 1, Enterococcus faecalis, Enterococcus faecium, Staphylococcus sciuri, Staphylococcus xylosus, Staphylococcus carnosus, Pseudomonas fluorescens, Burkholderia cepacia and Lactobacillus fructivorans. Is determined the capability to decarboxylate amino acids histidine, ornithine and tyrosine to 19 bacteria belonging to five groups of microorganisms.
|
El uso de materia prima de baja calidad, junto con la presencia de condiciones sanitarias inapropiadas durante el proceso, así como de una deficiente refrigeración en el producto terminado, contribuyen a la proliferación bacteriana (Márquez, García, 2007; Valladares, Faría, 2005) y por lo tanto a la formación de compuestos derivados de su metabolismo, tales como ácidos grasos, esteres, alcoholes, y compuestos nitrogenados como aminas biógenas (AB) (Zoecklein et al., 2001).
Las aminas biógenas son bases orgánicas producidas principalmente por descarboxilación microbiana de aminoácidos específicos que se encuentran en forma libre (Silla-Santos, 1996).
Las AB se encuentran en gran número en los alimentos y su concentración varía ampliamente; las más comunes son las monoaminas histamina (HI), tiramina (TY) y triptamina (TR) y las diaminas putrescina (PUT) y cadaverina (CAD). Estas son formadas a partir de los aminoácidos histidina, tirosina, triptófano, ornitina y lisina respectivamente (Landete et al., 2007b). Existen además poliaminas, como la espermina (SPM) y espermidina (SPD) que son derivadas únicamente de la putrescina.
Los alimentos que probablemente contienen los niveles más altos de estos compuestos son los productos lácteos, pescado y sus derivados, carne y sus derivados, vegetales fermentados, productos de la soya, y bebidas alcohólicas como el vino y la cerveza (Landete et al., 2007c), también pueden formarse en bajas concentraciones en alimentos no fermentados tales como frutas, vegetales, carne, leche y pescado, así como también durante el almacenamiento y el procesamiento de alimentos con calor (Önal, 2007).
Debido a la importancia de las aminas biógenas durante las últimas tres décadas, se han desarrollado métodos para la detección directa de estos compuestos, así como también de las bacterias que los producen. Los métodos más simples se basan en el crecimiento de los microorganismos en medios de cultivo diferenciales que indican la presencia de la amina por un incremento del pH. Existen además métodos enzimáticos, específicos para bacterias productoras de histamina, basados en la producción de peróxido de hidrógeno por la acción de la enzima oxidasa sobre la histamina (Sumner, Taylor, 1989).
Técnicas cromatográficas como cromatografía en capa fina (por sus siglas en inglés TLC) (García et al., 2005) y cromatografía líquida de alta presión (por sus siglas en inglés HPLC), también han sido aplicadas en la identificación y cuantificación de AB (González et al., 1998; Leitão et al., 2005; Loret et al., 2005; Contreras et al., 2007; Landete et al., 2007a). Más recientemente se ha extendido el uso de métodos moleculares como la reacción en cadena de la polimerasa (por sus siglas en inglés PCR) para la detección temprana y rápida de bacterias productoras de aminas biógenas, mediante la identificación de los genes que codifican la capacidad de descarboxilar aminoácidos (De las Rivas et al., 2006; Landeta et al., 2007; De las Rivas et al., 2007; Chen et al., 2008).
El estudio de la microbiota productora de aminas biógenas se realiza casi exclusivamente en quesos madurados. Se sabe muy poco del comportamiento de la microbiota presente en quesos blancos frescos en relación a la producción de aminas, por lo que este estudio pretende identificar algunas de las bacterias presentes en estos quesos y evaluar su efecto potencial frente a la producción de tres de las aminas más referenciadas (histamina, tiramina y putrescina). Por tanto el objetivo principal de este artículo es identificar bacterias potencialmente productoras de aminas biogenas aisladas de queso blanco fresco venezolano, por medio de los métodos de prueba de descarboxilación de aminoácidos y cromatografía en capa fina (TLC).
Con el fin de obtener un considerable número de aislados bacterianos, provenientes de queso blanco fresco dentro de los cuales se pudieran ubicar cepas potencialmente productoras de aminas biógenas, se realizó el aislamiento de algunos grupos de microorganismos que han sido reportados como productores de éstas en diferentes alimentos (Pseudomonas sp., coliformes, Staphylococcus sp., Enterococcus sp. y Bacterias ácido lácticas) de seis tipos de queso fresco (Mozzarella, Búfala, Cabra, Guayamano, Guayanés y Queso-Cuajada).
Los quesos analizados fueron adquiridos en supermercados de la ciudad, su peso aproximado fue de 500 g y su fecha de vencimiento osciló entre 10 y 30 posterior a la fecha de compra.
Todos los quesos fueron trasladados al laboratorio en frío, donde luego de su identificación fueron analizados en un lapso de tiempo inferior a las 24 h siguiendo el protocolo descrito en la norma COVENIN 938:83. Los análisis se realizaron por triplicado, correspondiente a la misma marca comercial y fueron adquiridos en los mismos supermercados.
Las muestras identificadas fueron procesadas según la norma COVENIN 1126:89. En condiciones de asepsia se separaron cinco porciones de diferentes partes del queso, de aproximadamente 10 g cada una, se cortaron en trozos pequeños y se mezclaron hasta obtener una muestra homogénea. A partir de esta se pesaron 10 g de muestra y se mezclaron con 90 mL de diluyente (citrato de sodio 2% p/v) (dilución 1:10) o con 10 mL del mismo (dilución 1:2), y a continuación fueron homogenizadas en un Stomacher Lab-Blender 400 por espacio de 1 min. Inmediatamente se tomó un 1 mL de la solución anterior y se llevó a un tubo de ensayo que contenía 9 mL de citrato de sodio (dilución 1:100) ó 1 mL del mismo diluyente (dilución1:4) y así se continuó sucesivamente hasta alcanzar las diluciones 1:1000 ó 1:8 según el caso (Banwart, 1995 modificado).
La capacidad de producción de histamina, tiramina y putrescina se estudió en los 20 aislados bacterianos mediante dos métodos: 1) prueba de descarboxilación de aminoácidos y 2) cromatografía en capa fina (TLC).
Esta prueba fue realizada siguiendo la metodología propuesta por Moeller (1955), incluida en el manual de pruebas bioquímicas de identificación de bacterias médicas de MacFaddin, (1976). El caldo Moeller (Merck 5437) base para descarboxilación fue preparado según las recomendaciones del fabricante, posteriormente fue suplementado al 1% con los aminoácidos precursores L-histidina (Sigma H8000), L-tirosina (Sigma T3754) y L-ornitina (Sigma O-2375), para luego ser distribuido y esterilizado en tubos de ensayo. Los tubos fueron inoculados con los aislados bacterianos utilizando un hisopo de madera estéril y cubiertos con 1 mL de parafina (Merck 7160) o aceite mineral estéril. La incubación se realizó a 37 °C durante 10 días. Junto a las 20 muestras se incubaron tres controles: a) medio de cultivo sin inóculo suplementado con aminoácido, b) medio de cultivo sin aminoácido con inóculo y c) medio de cultivo.
El método propuesto por García et al. (2005), fue utilizado para la determinación de los aislados productores de aminas biógenas.
Las cepas bacterianas se inocularon con la ayuda de un hisopo de madera estéril en caldo nutritivo (Merck 5443) y en caldo infusión cerebro corazón (Merck 10493) (ICC ó Brain Heart Infusion BHI) suplementado al 1% con el correspondiente aminoácido precursor, una vez inoculados los tubos se incubaron a 30 ºC por 3-7 días.
Adicionalmente se incubaron tres controles: a) medio de cultivo suplementado con aminoácido sin inóculo, b) medio de cultivo con inóculo sin aminoácido y c) medio de cultivo. Una vez concluido el tiempo de incubación, los tubos fueron centrifugados a 3000 rpm por 15 min y el sobrenadante fue almacenado a -20 °C hasta su uso.
Se prepararon soluciones estándar de las aminas histamina (Aldrich 271659), tiramina (Sigma T2879), y putrescina (Sigma P7505) al 2% en etanol al 40%(Sigma 459844).
En tubos eppendorf estériles de 1,5 mL se adicionaron 100 μL de la muestra o de la solución estándar de la amina, 10 μL de NaOH 4N (Sigma, 1310-73-2), 100 μL de fosfato disódico 250 mM (pH 9) estéril y previamente refrigerado (MercK 106370), y 200 μL de solución de cloruro de dansilo (50 mg de cloruro de dansilo Sigma D2625 en 10 mL de acetona, Sigma 179124). Esta mezcla se incubó en oscuridad a 55 ºC por 1 h. Transcurrido este tiempo se dejó enfriar y se almacenó a -20 ºC hasta su uso.
Los estándares de las aminas y las muestras derivatizadas fueron fraccionadas en placas de sílica gel 60 F254 (20 x 20) (Merck 1.05548.0001) de la siguiente forma: a 2 cm del borde inferior de la placa se aplicó con un capilar 10 μL de la solución estándar de la amina, 10 μL de cada una de las muestras y 10 μL de los controles, con una distancia entre marcas de 1,2 cm. Una vez preparada la placa se introdujo en la cámara cromatografía que contenía la mezcla de elución o fase móvil, constituida por cloroformo (Sigma 472476)-trietilamina (Sigma 90337) (4:1), dicha cámara permaneció en una campana de extracción de gases mientras se desarrolló la separación (entre 2 y 3 h).
Cuando el frente de la corrida ascendió hasta alcanzar aproximadamente 2 cm del borde superior de la placa, la cromatografía fue interrumpida y las placas se dejaron secar en campana de extracción de gases por aproximadamente 30 min, luego de los cuales se rociaron con solución reveladora compuesta por isopropanol (Sigma 673773) / trietanolamina (Sigma T1377) (8:2) a 15-20 cm de distancia y se dejaron secar para luego ser observadas bajo luz UV (360 nm). Visualizados los spots o manchas fluorescentes bajo luz UV se marcaron con un lápiz, para posteriormente calcular los valores de Rf y Rx.
Para el análisis estadístico de los resultados se aplicó un análisis de varianza (ANOVA) multifactorial con interacción de segundo orden, considerando un nivel de significancia de α= 0,05 para evaluar si existían diferencias entre la concentración de bacterias determinadas por cada tipo de queso. También fueron evaluadas las diferencias existentes entre los resultados observados en el en dos periodos de tiempos mediante la prueba de rangos múltiples de Duncan. Para la validez de los resultados se comprobaron los supuestos de normalidad, homogeneidad de varianza y aleatoriedad.
Para el análisis de los parámetros físicos y químicos (pH, aw, acidez y cloruros) se realizó un ANOVA de una sola vía, con un nivel de significancia de α =0,05.
La microbiota obtenida tanto en análisis de los seis tipos de queso en los dos periodos, como la que fue positiva a la producción de aminas biógenas fue comparada sobre la base de características fenotípicas y bioquímicas, aplicando un análisis multivariado con el programa MiniTab versión 15, y expresando los resultados gráficamente mediante dendogramas.
Grupos de microorganismos presentes en el queso blanco fresco La evaluación microbiológica de seis tipos de queso blanco fresco, en los dos períodos de tiempo tuvo como objetivo el aislamiento de un considerable número de cepas bacterianas que pudieran ser productoras de AB. Se trabajó con diluciones bajas, por lo cual se obtuvieron recuentos elevados. Se evaluó el recuento de Pseudomonas sp., Staphylococcus sp., Enterococcus sp., BAL y coliformes con miras a aislar diversas especies comúnmente asociadas con la producción de aminas biógenas como la histamina, tiramina y putrescina (Chen et al., 1989; González et al., 1998; Martuscelli et al., 2005; Landete et al., 2007a; De las Rivas et al., 2008).
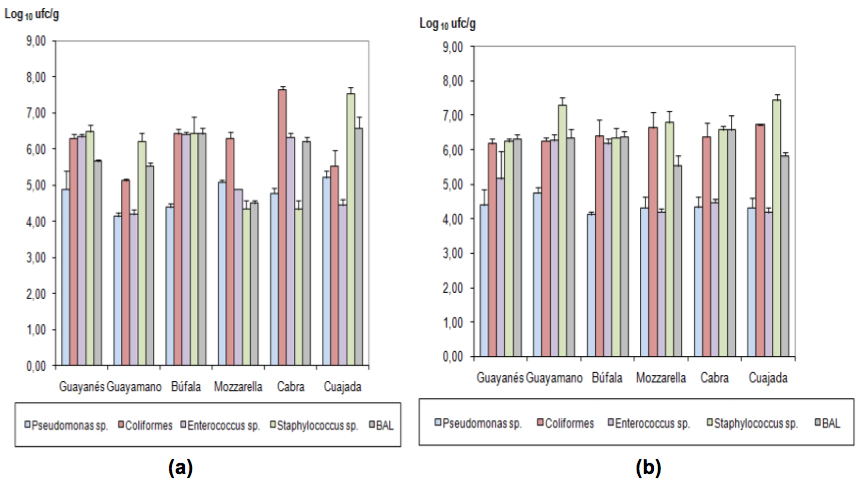
Figura 1 – Microorganismos presentes en quesos blancos frescos
“a” periodo mayo-junio de 2008, “b” periodo febrero-marzo 2009
Fonte: Elaborado pelos autores (2016)
En la Figura 1 se observan los resultados del recuento de los grupos de microorganismos evaluados, de estos se puede destacar que el género Pseudomonas presentó los menores recuentos en la mayoría de los quesos analizados. En el primer periodo evaluado los recuentos más bajos se observaron en el queso guayamano (1,4x104 ufc/g) y los más altos en queso cuajada (1,7x105 ufc/g), mientras que en el 2009, los menores recuentos de Pseudomonas sp., fueron los de queso de búfala (1,3x104 ufc/g) y los más altos los de queso guayamano (6,0x104 ufc/g). El queso de cabra fue el que presentó la mayor diferencia en el número de coliformes entre los dos periodos de tiempo Figura 1 (b), registrando recuentos de 4,4x107 y 3,2x106 en los periodos 2008 y 2009 respectivamente. En los quesos restantes el número de ufc/g de coliformes osciló en el orden de 1,5x106 y 6,6x106. Autores como Maldonado y Llanca (2008) reportaron valores promedios para coliformes totales en queso “de mano” de 2,2x106 NMP/g, los cuales superaron el valor máximo recomendado por la norma COVENIN 3821:2003 (2,1x104 NMP/g), mientras que Díaz y González (2001), hallaron contenidos de coliformes totales superiores a 1,0x10 4 NMP/g en muestras de queso blanco fresco semi-duro. Araya et al. (2008) encontró valores comprendidos entre < 10 ufc/g y 1,3x106 ufc/g para coliformes totales en muestras de queso de cabra elaborados con leche pasteurizada.
El recuento de Enterococcus sp. para los seis tipos de queso en los dos periodos de tiempo osciló entre 1,5x104 ufc/g y 2,5x106 ufc/g, los cuales son inferiores a los reportados por Ferrer et al. (1987), en queso tipo palmita elaborado con leche cruda en el que se alcanzaron valores de 1,0x107 ufc/g.
Los valores más altos del recuento de Staphylococcus sp. se presentaron en el queso-cuajada, con 3,6x107 y 2,9x107 ufc/g en 2008 y 2009 respectivamente, mientras que los menores recuentos se exhibieron en queso mozzarella y cabra, en un orden de magnitud de 2,4x104 ufc/g en ambos casos. En queso “de mano” Maldonado y Llanca (2008), reportaron valores promedio de 2,9x106 ufc/g de Staphylococcus spp., mientras que en queso blanco fresco semiduro Díaz y González (2001), reportan contenidos de S. aureus de 1,5x105 ufc/g.
En cuanto al contenido de Bacterias ácido lácticas (BAL), los menores recuentos que fueron obtenidos en queso mozzarella con 3,2x104 y 3,9x105 ufc/g. Los valores más altos se presentaron en la cuajada, mientras que se encontró un mayor número de bacterias ácidos lácticas en el queso de cabra (Figura 1). El recuento general de las BAL en los seis tipos de queso estuvo comprendido entre 3,2x104 y 4,9x106 . Ferrer et al. (1987), reportan en queso tipo palmita recuentos de 1x109 ufc/g de Lactobacillus sp.
En cuanto a la comparación estadística de los datos, el análisis de varianza para el recuento (ufc/g) de Pseudomonas sp. determinó que los factores tipo de queso y periodo tienen efecto sobre el contenido de Pseudomonas sp., sin embargo es la interacción de estos dos factores la que presenta un mayor efecto (P=0,0000). El gráfico de interacción que se presenta en la Figura 2 (a) señala que los quesos cuajada, mozzarella, y cabra presentaron un recuento de Pseudomonas sp., superior en el periodo mayo-junio de 2008.
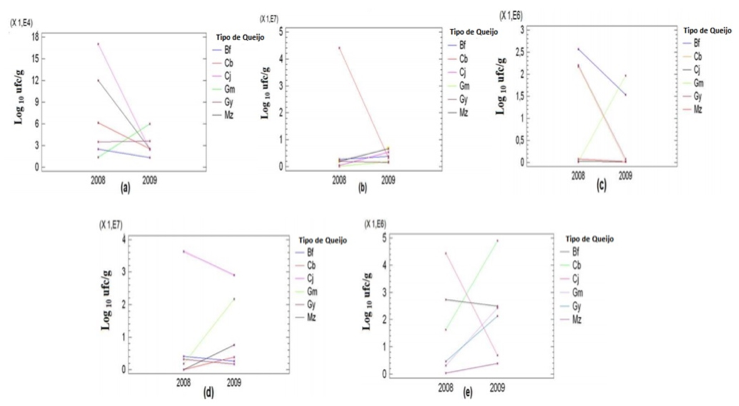
Figura 2 – Gráficos de interacción del tipo de queso y periodo sobre el recuento en Log (ufc/g)
de los grupos de microorganismos evaluados a) Pseudomonas sp., b) coliformes,
c) Enterococcus sp., d) Staphylococcus sp. y e) BAL
Fonte: Elaborado pelos autores (2016)
Contrario a lo encontrado en el queso guayamano; en el queso guayanés por su parte, no se establecieron diferencias significativas en el recuento en los dos periodos de tiempo. El gráfico de interacción para el recuento de coliformes Figura 2 (b) reveló que se presentaron diferencias estadísticamente significativas (P=0,0000) entre el queso de cabra y los cinco quesos restantes en el periodo de estudio.
El ANOVA empleado en el análisis de los resultados del recuento de Enterococcus sp. determinó que la interacción de los factores tipo de queso y periodo posee un efecto estadísticamente significativo (P=0,0000) en el recuento de este grupo de bacterias. El gráfico de interacción que se observa en la Figura 2 (c) exhibe diferencias estadísticamente significativas entre el queso de búfala y los quesos guayamano, mozzarella y cuajada en el periodo mayo-junio de 2008, mientras que en el periodo febrero-marzo de 2009 el queso guayamano presentó el mayor recuento de Enterococcus sp. y junto al queso de búfala presentaron diferencias estadísticamente significativas, respecto a los demás quesos analizados.
En el recuento de Staphylococcus sp. se presentaron diferencias estadísticamente significativas (P=0,0274) entre la cuajada y los demás tipos de queso en los dos periodos de tiempo, los otros quesos no presentaron diferencias estadísticamente significativas (P=0,1271) en el recuento de Staphylococcus sp., en los dos periodos de tiempo, estos resultados se presentan en la Figura 2 (d).
El análisis de varianza del recuento de bacterias ácido lácticas en los seis tipos de queso determinó que existe un efecto del factor tipo de queso sobre el recuento de las BAL, mientras que el factor periodo no mostró efecto estadísticamente significativo (0,2898); sin embargo la interacción de los dos factores si presentó un efecto significativo (P=0,0147) sobre el recuento en ufc/g de este grupo bacteriano. El queso mozzarella fue el único que no presentó diferencias significativas (P=0,2898) en el recuento de BAL en los dos periodos de tiempo.
De manera global, los valores promedio del contenido de microorganismos fueron elevados (>104 ufc/g) en todos los quesos en los dos periodos de tiempo, lo que podría indicar el empleo de una materia prima de baja calidad microbiológica, o una deficiencia de los sistemas de 55 higiene utilizados en la elaboración de queso blanco fresco, sin embargo, Ferrer et al. (1987), señalan que los altos recuentos de coliformes, Enterococcus sp., y Lactobacillus sp., encontrados en queso tipo palmita elaborado con leche sin pasteurizar sugieren un importante papel de estos microorganismos en la determinación de las propiedades organolépticas del mismo.
Identificación de los aislados como resultado del análisis microbiológico de los seis quesos bajo estudio en los dos periodos de tiempo evaluados, se seleccionaron 19 aislados bacterianos, los cuales se agruparon según la similitud de sus características morfológicas macroscópicas observadas sobre medios de cultivo diferenciales.
3.1. Evaluación de la capacidad de producción de aminas biógenas por los aislados bacterianos
Una vez identificados los diecinueve aislados se evaluó la capacidad para descarboxilar los aminoácidos histidina, tirosina u ornitina y producir la subsecuente amina biógena, histamina, tiramina o putrescina, mediante la determinación de la descarboxilación de aminoácidos en medios de cultivos líquidos y por cromatografía en capa fina (TLC).
3.2. Determinación de la descarboxilación de aminoácidos mediante siembra en medios de cultivo líquidos
La descarboxilación de un aminoácido producto del metabolismo microbiano, se evidencia por un cambio de color del púrpura de bromocresol consecuencia del aumento del pH cuando se usa medio Moeller Figura 3, se acepta como resultado positivo el viraje de amarillo a púrpura intenso desarrollado a lo largo del tubo de ensayo luego de 15 días de incubación como máximo.

Figura 3 – Prueba de descarboxilación de aminoácidos
Fonte: Elaborado pelos autores (2016)
Panel A: Prueba negativa a la descarboxilación de aminoácidos (a) muestra, (b) control 1: medio de cultivo, (c) control 2: medio de cultivo sin inóculo y con aminoácido, (d) control 3: medio de cultivo con inóculo y sin aminoácido. Panel B: Prueba positiva a la descarboxilación de aminoácidos, (a) muestra, (b) control 1: medio de cultivo, (c) control 2: medio de cultivo con inóculo y sin aminoácido, (d) control 3: medio de cultivo sin inóculo y con aminoácido.
Los aminoácidos como la histidina, tirosina y ornitina entre otros, pueden ser metabolizados por diversos grupos bacterianos que han sido descritos como potenciales formadores de AB. De forma muy general, las enterobacterias se asocian con la producción de cadaverina y/o histamina, mientras que la microbiota láctica (especialmente los Enterococcus) suele ser productora de tiramina (Bover et al., 2001). En la Tabla 1 se presentan los resultados de la prueba de descarboxilación de aminoácidos. Por este método ninguno, resultó positivo a la descarboxilación de histidina, observándose solo actividad frente a tirosina y ornitina.
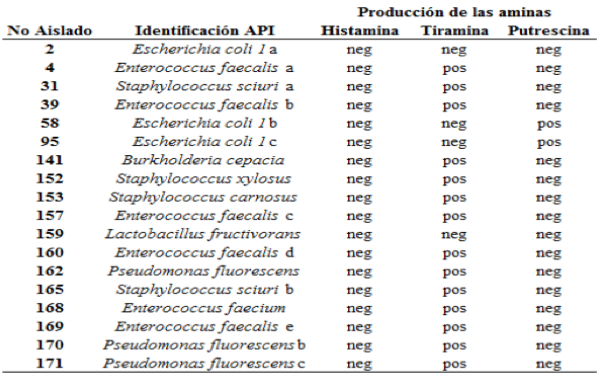
Tabla 1 – Aislados productores de aminas biógenas por la prueba de descarboxilación de aminoácidos
Fonte: Elaborado pelos autores (2016)
Los aislados identificados como E. coli 1b (proveniente de queso de cabra 2008) y E. coli 1c (proveniente de queso guayamano 2008) fueron positivos a la descarboxilación de ornitina, dicho resultado es consecuente con lo expuesto por Landete et al. (2007b), mientras que E. coli 1a (proveniente de queso cuajada 2008), no evidenció capacidad para descarboxilar ninguno de los tres aminoácidos junto con Lactobacillus fructivorans (proveniente de queso de búfala 2009), este comportamiento pudiera estar explicado en el hecho de que la capacidad para formar una o más aminas, así como la intensidad aminogénica varía ampliamente entre especies e incluso entre cepas bacterianas y puede modificarse según las condiciones ambientales en las que éstas se desarrollen (Bover et al., 2001).
Lactobacillus fructivorans fue estudiado por Moreno et al. (2003), quienes no determinaron actividad para descarboxilar ninguno de los tres aminoácidos aquí evaluados (histidina, tirosina, y ornitina) y sólo observaron capacidad para descarboxilar arginina en una cepa.
Quince aislados descarboxilaron tirosina para producir tiramina, seis de ellos identificados como Enterococcus faecalis (diferenciados con letras minúsculas según provinieran de los quesos: “a” para queso de búfala 2008, “b” cabra 2008, “c” búfala 2009, “d” mozzarella 2009, “e” cabra 2009 y “f” guayamano 2009) y uno identificado como Enterococcus faecium proveniente de queso guayanés del periodo 2009. Estos resultados no difieren de los reportados por Sánchez et al. (2009), quienes estudiaron la capacidad de descarboxilar aminoácidos en cepas de E. faecalis, encontrando que una cepa de E. faecalis mostró una débil actividad descarboxilasa de ornitina, mientras que cinco de nueve cepas estudiadas mostraron capacidad para descarboxilar tirosina. Este último aminoácido es el más reportado en la descarboxilación por Enterococcus sp. (Landete et al., 2007b; Komprda et al., 2008; Ogier y Serror, 2008). En el mismo estudio Sánchez et al. (2009), encontraron que cepas de E. faecium demostraron una mayor capacidad de descarboxilar los aminoácidos tirosina, ornitina, lisina e histidina.
Adicionalmente los aislados identificados como Staphylococcus sciuri a (procedente de queso búfala 2008), Staphylococcus sciuri b (proveniente de queso guayanés 2009), Staphylococcus carnosus y Staphylococcus xylosus procedentes ambos de queso guayamano 2009, también fueron positivos a la descarboxilación de tirosina, dichos resultados concuerdan con los obtenidos por De las Rivas et al. (2008), quienes luego de determinar aminas biógenas en chorizo seco curado encontraron que las bacterias con capacidad descarboxilante de tirosina y en consecuencia productoras de tiramina pertenecían principalmente a cepas de S. carnosus, los cuales simultáneamente produjeron β-feniletilamina. Kung et al. (2008), hallaron cepas de S. carnosus productoras de histamina, cadaverina y feniletilamina en productos de pescado y no detectaron presencia de tiramina producida por dichas bacterias. Hsu et al. (2009), evaluaron bacterias productoras de histamina en caldo soya tripticasa suplementado al 1% con histidina, encontrando que diez de cuarenta cepas evaluadas correspondieron a S. xylosus y una a S. sciuri, las cuales produjeron 51,4 y 6,0 ppm de histamina respectivamente.
Por otra parte existen estudios como los de Yongjin et al. (2007), quienes afirman que asociaciones de S. carnosus, S. xylosus y L. sakei tienen un efecto amino-negativo, que puede prevenir la formación de aminas biógenas, o reducir en gran medida (90%) la presencia de putrescina, cadaverina, y tiramina en salsas de soya. Los tres aislados identificados como Pseudomonas fluorescens a, b y c según hubiesen sido obtenidos de los quesos guayanés 2009, guayamano 2009 y guayanés 2009 respectivamente, además de Burkholderia cepacia obtenida de queso búfala 2009, también resultaron positivas a la descarboxilación de tirosina. Martuscelli et al. (2005), determinaron mediante el uso de un medio de cultivo la descarboxilación de aminoácidos, observando que diferentes especies de Pseudomonas presentaban cambios de coloración en el medio, característica que suponía la formación de las aminas histamina, tiramina, triptamina, feniletilamina, cadaverina, y putrescina entre otras.
En dicho estudio las cepas de Pseudomonas evaluadas descarboxilaron todos los aminoácidos precursores estudiados. Por su parte Chen et al. (2008), observaron descarboxilación de histidina por cepas de Pseudomonas fluorescens. No existen reportes de la descarboxilación de aminoácidos con la subsecuente formación de aminas biógenas por cepas de Burkholderia cepacia.
3.3. Evaluación de la capacidad de producir aminas biógenas por cromatografía en capa fina (TLC)
Inicialmente, durante los ensayos de estandarización de la técnica, se realizó una cromatografía con los patrones, en la que se determinaron los valores de factor de retención (Rf), (Walton y Reyes, 1975). Para histamina (0,890), tiramina (0,775) y putrescina (0,644), conforme Figura 4. En esta también se pudo establecer que al inocular 10 μL de solución patrón de la amina se obtenían marcas fácilmente diferenciables, sin observar la aparición de marcas que interfirieran en la lectura de los patrones. El color de las marcas de los patrones bajo luz UV fue naranja amarillento suave para histamina, y verde azulado para tiramina y putrescina.

Figura 4 – Separación de los patrones de histamina, tiramina y putrescina por TLC
Fonte: Elaborado pelos autores (2016)
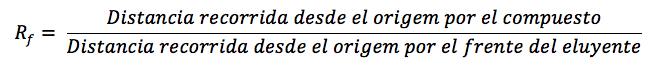
García et al. (2005), utilizaron TLC para detectar simultáneamente dos tipos de aminas producidas por las cepas bacterianas E. faecium BIFI-58 y Lactobacillus 30a, la primera productora de feniletilamina y tiramina, y la segunda productora de histamina y putrescina. Para ambas bacterias se pudo observar que las marcas características de cada amina eran claramente diferenciables. Estos autores reportaron valores de Rf de 0,849 para histamina, 0,915 para tiramina y 0,458 para putrescina, además de coloraciones características para cada una de las aminas: verde azulado para putrescina, tiramina y feniletilamina, y naranja amarillento para histamina.
Los valores Rf obtenidos en este estudio difieren de los encontrados por otros autores, dicha variabilidad podría estar asociada al tipo y proporción de la fase móvil, a la saturación de la cámara de desarrollo y en general a dos procesos que influyen en cualquier tipo de cromatografía: la migración de las zonas y el ensanchamiento de las zonas. Respecto a la migración de las zonas, se debe considerar que inicialmente el sustrato se halla concentrado en una zona muy estrecha y bien definida en la base de la placa (fase estacionaria) y, que al entrar en contacto con la fase móvil, sus moléculas se distribuyen entre las dos fases, se mueven rápidamente pasando y volviendo a pasar de una fase a otra.
A medida que pasa el disolvente el sustrato se desplaza y los bordes de la zona se vuelven difusos, algunas moléculas del sustrato se desplazan más rápidamente que el promedio de ellas, mientras que otras lo hacen más lentamente porque durante una parte de tiempo permanecen en el interior de la fase estacionaria o absorbida en la superficie de ésta. Cuanto más atraídas por la fase estacionaria tanto más lentamente avanzan por la placa.
En cuanto al ensanchamiento de las zonas, una pequeña cantidad de sustrato colocada en la base de la placa cromatografía, es retenida por ésta formando inicialmente un disco delgado, es decir, una zona estrecha y de bordes muy definidos, extendida por toda la sección transversal de la placa, a medida que se desplaza a lo largo de la placa, esta zona se ensancha y se vuelve más difusa, básicamente debido a la difusión turbulenta, pues las moléculas del sustrato no siguen necesariamente el camino más directo entre los gránulos de la fase fija. Cuando encuentran un gránulo cada una de ellas lo rodea de forma aleatoria, lo que genera un retraso en el ascenso por la placa de algunas de ellas, dicho movimiento aleatorio da lugar al ensanchamiento de las zonas (Walton y Reyes, 1978). Por estos motivos es indispensable realizar el estudio de migración de los patrones de las aminas cada vez que se estandariza o realiza una evaluación de compuestos orgánicos por TLC.
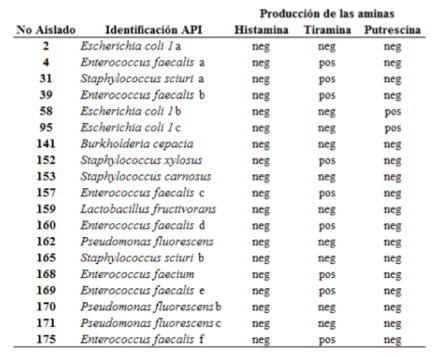
Tabla 2 – Aislados productores aminas biógenas por cromatografía en capa fina
Fonte: Elaborado pelos autores (2016)
En relación a los resultados observados por el método de descarboxilación de aminoácidos y por cromatografía en capa fina se presentaron diferencias en el número de aislados positivos a la producción de tiramina, trece aislados presentaron el mismo comportamiento por ambos métodos (9 positivos a la producción de tiramina, 2 positivos a la producción de putrescina y dos no productores por ningún método).
Dichas diferencias entre los métodos pueden estar sustentadas en el hecho de que los medios diferenciales que contienen un indicador de pH, al parecer describen reacciones de falsos positivos debido a la formación de otros compuestos alcalinos y/o a periodos de incubación escasos (Chen et al., 1989; Moreno et al., 2003), al igual que falsos negativos, probablemente ocasionados por el bajo pH inicial del medio, que puede excluir el crecimiento de algunas bacterias productoras de aminas biógenas (Niven et al., 1981).
En los seis tipos de queso se detectó la presencia de coliformes, Enterococcus sp., Staphylococcus sp., Pseudomonas sp., y bacterias ácido lácticas. Siendo Staphylococcus sp., coliformes y Enterococcus sp., la población dominante.
De las 19 bacterias seleccionadas para evaluar la producción de aminas biógenas Enterococcus faecalis representó el 32%, E. coli 1 y Pseudomonas fluorescens el 16%, Staphylococcus sciuri el 10,5% y el 21,5% restante estuvo integrado por Burkholderia cepacia, Staphylococcus xylosus, Staphylococcus carnosus y Lactobacillus fructivorans.
Los valores de los parámetros físicos y químicos analizados se encontraron en un rango que favorece el crecimiento de una gran variedad de bacterias por lo que no fue posible correlacionar un parámetro específico con la aparición de una bacteria en particular.
La prueba de descarboxilación de aminoácidos en medio Moeller indicó que el 80% de las cepas bajo estudio producían tiramina, mientras que por la técnica de cromatografía en capa fina tan solo un 47% de los aislados resultaron positivos. Por ambos métodos 10% de las cepas produjeron putrescina y ninguna fue productora de histamina.
Araya V, Gallo L, Quesada C, Chaves C, Arias M. (2008); Evaluación bacteriológica de la leche y queso de cabra distribuidos en el área metropolitana de San José, Costa Rica. ALAN 58:20- 6.
Bover S, Hugas M, Izquierdo M, Vidal M. (2001); Amino acid-decarboxylase activity of bacteria isolated from fermented pork sausages. Int J Food Microbiol 66:185-9
Contreras M, Izquierdo P, Allara M, García A, Torres G, Céspedes E. (2007); Determinación de aminas biógenas en quesos madurados. Rev Cient Fac Cs Vets Uni Zulia [serie en línea]. 17:1- 4. Disponible en (http://www.scielo.com). Consultado Abr 24, 2010.
De las Rivas B, Marcobal A, Carrascosa A, Muñoz R. (2006); PCR Detection of foodborne bacteria producing the biogenic amines histamine, tyramine, putrescine and cadaverine. J Food Prot 69:2509-14.
De las Rivas B, Capillas C, Carrascosa A, Curiel J, Jiménez F, Muñoz R. (2008); Biogenic amine production by gram-positive bacteria isolated from spanish dry-cured “chorizo” sausage treated with high pressure and kept in chilled storage. Meat Sci 80:272-7.
Ferrer A, Urdaneta D, Rincón Z. (1987); Evaluación fisicoquímica y microbiológica del queso tipo palmita venezolano. Rev Cient Uni Andes. 4:133-47.
González D, Cuesta P, Rodríguez A. (1998); Biogenic amine production by wild lactococcal and leuconostoc strains. Lett Appl Microbiol 26:270-4.
Hsu H, Chuang T, Lin H, Huang Y, Lin C, Kung H, Tsai Y. (2009); Histamine content and histamine-forming bacteria in dried milkfish (chanos chanos) products. Food Chem 114:933-8.
Komprda T, Burdychová R, Dohnal V, Cwiková O, Sládková P, Dvořáčková H. (2008); Tyramine production in Dutch-type semi-hard cheese from two different producers. Food Microbiol 25:219-27.
Kung H, Chien L, Liao H, Lin C, Liaw E, Chen W, Tsai Y. (2008); Chemical characterization and histamine-forming bacteria in salted mullet roe products. Food Chem 110:480-5.
Landete J. De las Rivas B, Marcobal A. Muñoz R. (2007a); Molecular method for detection of biogenic amine-producing bacteria on food. Int. J. Food Microbiol 18: 258-69
Landete J, Ferrer S, Pardo I. (2007b); Biogenic amine production by lactic acid bacteria, acetic bacteria and yeast isolated from wine. Food Contr 18:1569-74.
Landete J, Pardo I, Ferrer S. (2007c); Tyramine and phenylethylamine production among lactic acid bacteria isolated from wine. Int J Food Microbiol 115:364-8.
Leitão M, Marques A, San Romão M. (2005); A survey of biogenic amines in commercial portuguese wines. Food Contr 16:199-04.
Loret S, Deloyer P, Dandrifosse G. (2005); Levels of biogenic amines as a measure of the quality of the beer fermentation process: data from Belgian samples. Food Chem 89:519-25.
Maldonado R, Llanca L. (2008); Estudio de la calidad del queso de mano comercializado en el municipio de Girardot, estado Aragua, Venezuela. Rev Cient Fac Agron UCV [en línea]. 18:4- 9. Disponible en Scielo (http://www.scielo.mhtml). Consultado May 7, 2008.
MacFaddin JF. (1976); Biochemical tests for identification of medical bacteria. Baltimore, USA: The Williams & Wilkins Company. p. 12-5, 27-32, 52-64, 75-79.
Martuscelli M, Gardini F, Torriani S, Mastrocola D, Serio A, Chavez L, Schirone M, Suzzi G. (2005); Production of biogenic amines during the ripening of pecorino abruzzese cheese. Int Dairy J 15:571-8.
Moreno M, Polo M, Jorganes F, Muñoz R. (2003); Screening of biogenic amine production by lactic acid bacteria isolated from grape must and wine. Int J Food Microbiol 84:117-23.
Niven C, Jeffrey M, Corlett D. (1981); Differential plating medium for quantitative detection of histamine-producing bacteria. Appl Environ Microbial 41:321-2.
Norma COVENIN 938. (1983); Leche y productos lácteos. Métodos para la toma de muestra. Comisión Venezolana de Normas Industriales. Caracas, Venezuela: FONDONORMA.
Norma COVENIN 1126. (1989); Alimentos. Identificación y preparación de muestras para el análisis microbiológico. 1ra revisión. Comisión Venezolana de Normas Industriales. Caracas, Venezuela: FONDONORMA.
Norma COVENIN 3821. (2003); Queso blanco. Comisión Venezolana de Normas Industriales. Caracas, Venezuela: FONDONORMA.
Ogier JC, Serror P. (2008); Safety assessment of dairy microorganism: The Enterococcus genus. Int J Food Microbiol 126:291-01.
Önal A. (2007); A review: Current analytical methods for determination of biogenic amines in foods. Food Chem 103:1475-86.
Sánchez A, Bem N, Abriouel H, López R, Veljovic K, Martinez M, Ljubisa M, Gálvez A. (2009); Virulence factors, antibiotic resistance, and bacteriocins in enterococci from artisan foods of animal origin. Food Contr 20:381-5.
Silla-Santos M. (1996); Biogenic amines: their importance in foods. Int J Food Microbiol 29:213-31.
Sumner S, Taylor S. (1989); Detection method for histamine-producing, dairy-related bacteria using diamine oxidase and leucocrystal violet. J Food Prot 52:105-6.
Valladares O, Faría J. (2005); Propuestas para mejorar la industria quesera en Venezuela. Manual de Ganadería Doble Propósito, Rev Cient Uni Zulia 34:21-35.
Walton H, Reyes J. (1978); Análisis químico e instrumental. Zaragoza España: Acribia p. 270- 290.
Yongjin H, Wenshui X, Xiaoyong L. (2007); Changes in biogenic amines in fermented silver carp sausages inoculated with mixed starter cultures. Food Chem 104:188-95.
Zoecklein B, Fugelsang K, Gump B, Nury F. (2001); Análisis y producción de vino. Zaragoza España: Acribia, S.A. p. 159-61.
1. Universidad Simón Bolívar (USB), Programa de Maestría en Ciencias de los Alimentos, Sartenejas – Caracas – Miranda – Venezuela. Email: albenavides@uniboyaca.edu.co
2. Universidad Tecnológica Federal do Paraná (UTFPR), Programa de Maestría en Ingeniería de Producción, Ponta Grossa – Paraná – Brasil. Email: ingdanielher@gmail.com
3. Universidad Tecnológica Federal do Paraná (UTFPR), Programa de Maestría en Ingeniería de Producción, Ponta Grossa – Paraná – Brasil. Email: hugo_868@hotmail.com
4. Universidad Tecnológica Federal do Paraná (UTFPR), Programa de Maestría en Ingeniería de Producción, Ponta Grossa – Paraná – Brasil. Email: guilherme.ribeiro91@hotmail.com
5. Universidad Estadual de Maringá (UEM), Programa de Maestría en Ingeniería de Alimentos, Maringá – Paraná – Brasil. Email: gusmatias@gmail.com
6. Universidad Federal do Rio Grande do Sul (UFRGS), Programa de Maestría en Ingeniería de Producción, Porto Alegre – Rio Grande do Sul – Brasil. Email: fernando-lermen@hotmail.com
7. Universidade Tecnológica Federal do Paraná (UTFPR), Ponta Grossa – Paraná – Brasil. Email: leocir3d@hotmail.com